Dans ce nouveau chapitre de la rubrique Du côté du laboratoire, nous abordons la place de Pseudomonas aeruginosa en tant qu’agent responsable d’infections et d’épidémies associées aux soins, sa surveillance et sa détection dans l’environnement hospitalier et dans le cadre d’investigations, l’analyse comparative de souches cliniques et environnementales en cas d’épidémie, et les mesures de prévention.
Introduction
P. aeruginosa (PA), également appelé bacille pyocyanique en raison des pigments bleutés qu’il peut produire, est une bactérie pathogène opportuniste à l’origine d’infections variées, aussi bien aiguës (pneumonies, infections urinaires, bactériémies…), principalement chez les sujets immunodéprimés ou présentant une affection maligne [1], que chroniques, notamment dans le cadre de la mucoviscidose. Quatrième agent étiologique responsable d’IAS en France, il représente une source majeure d’IAS, avec une situation endémique dans les unités de soins intensifs et de réanimation, où il est responsable de presque 20% des infections nosocomiales (données Réa-Raisin1 et Santé Publique France 2018, enquête nationale de prévalence 2022). En plus d’une résistance naturelle à de nombreux antibiotiques utilisés en population générale, PA possède de nombreux mécanismes acquis de résistance aux antibiotiques, avec l’émergence de phénotypes multirésistants (MDR [multidrug resistant]) ou ultrarésistants (XDR) responsables de situations cliniques complexes pouvant aboutir à des impasses thérapeutiques et à une morbi-mortalité considérable [2]. PA est classé parmi les pathogènes ESKAPE2 [3], et les souches productrices de carbapénémase figurent dans le groupe des agents pathogènes prioritaires de haut niveau d’après l’Organisation mondiale de la santé3. L’espèce PA est considérée comme de diffusion non clonale, mais certaines lignées ou génotypes de PA ont émergé grâce à leurs capacités adaptatives particulières [4], et sont particulièrement répandus dans l’environnement hospitalier en raison de leur pouvoir de persistance [5,6] qui leur confère un succès dans les IAS et les épidémies. Ces génotypes de PA sont qualifiés de clones à haut risque épidémique (EHR4) [7]. Après une première partie détaillant les caractéristiques et le cycle épidémiologique de PA, nous aborderons ses méthodes de détection dans l’environnement avant de parler des outils de typage moléculaire nécessaires aux investigations d’épidémie. Enfin, nous détaillerons les principales mesures de prévention des IAS et des épidémies liées à PA en milieu de soins.
Un pathogène nosocomial lié à l’eau
PA, bacille à coloration de Gram négative (BGN) non fermentant, de la famille des γ-protéobactéries, est une bactérie environnementale dite ubiquitaire, capable de coloniser un large éventail de biotopes terrestres, hydriques, anthropisés ou non [8]. PA constitue une menace grandissante dans les IAS du fait de sa capacité à développer des mécanismes de résistance aux antibiotiques, notamment par le biais de mutations chromosomiques, de surexpression de gènes naturels (céphalosporinases, pompes d’efflux…), de transferts horizontaux de gènes de résistance (incluant des carbapénémases et des bêtalactamases à spectre étendu), et de la propension à l’expansion clonale de génotypes EHR, souvent multirésistants aux antibiotiques [7]. Dans son rapport de 20225, le Centre national de référence de la résistance aux antibiotiques faisait état d’une augmentation de la proportion de souches de PA productrices de bêtalactamases à spectre étendu ou carbapénémases (+14,3%), avec une prédominance des carbapénémases de type VIM-26 et NDM-17. PA est qualifié d’opportunistic premise plumbing pathogen (OPPP) en raison de sa facilité à persister dans les réseaux de plomberie et les surfaces humides, de sa versatilité métabolique, de sa résistance aux agents antimicrobiens (antibiotiques, mais aussi antiseptiques et désinfectants) et de sa tolérance vis-à-vis de certains métaux comme le cuivre, utilisé classiquement pour la conception des réseaux d’eau hospitaliers [9]. Ainsi, les réseaux d’eau des établissements de santé constituent des niches technologiques fréquemment colonisées par PA [10], réservoirs de souches opportunistes exposant les patients à un risque d’IAS. Le cycle épidémiologique de PA dans les IAS est complexe et l’environnement hospitalier agit comme un réservoir et un amplificateur de PA [11,12] (Figure 1). La quantification de ce risque est délicate en raison des nombreux échanges qui s’opèrent entre les deux compartiments (hommes et environnement). Il est parfois difficile d’expliquer la voie exacte de transmission et d’affirmer la chronologie de la transmission environnement-patient. Néanmoins, la présence de PA dans l’eau du réseau étant clairement identifiée comme un facteur de risque d’infection à PA, notamment dans les unités accueillant des patients à risque tels que les patients immunodéprimés, un des moyens de lutte efficaces consiste à mettre en place des mesures de prévention et de contrôle de la biocontamination environnementale [13].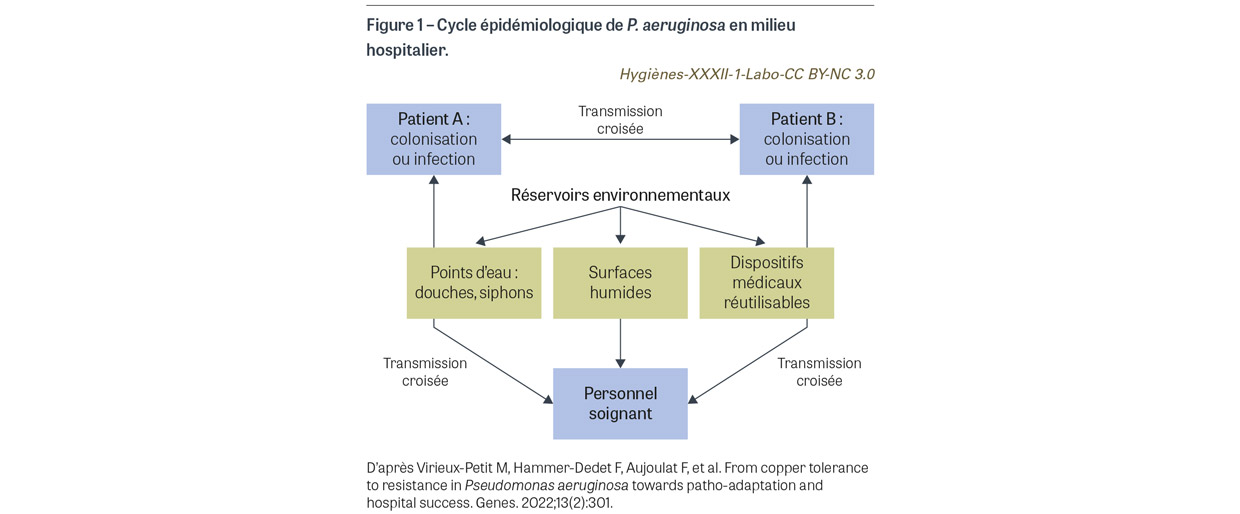
L’environnement hospitalier en tant que source de contamination et de transmission
Au sein des services de soins, PA est régulièrement isolé au niveau des points d’eau (siphons et brise-jet), notamment ceux utilisés pour le lavage des mains, la toilette des patients ou l’entretien des locaux [14,15]. Les siphons constituent des niches technologiques parfaites pour PA, à l’interface entre la rétro-colonisation à partir des canalisations et les activités anthropiques. Ils constituent ainsi des sites privilégiés de colonisation et de persistance de PA, des réservoirs de souches responsables d’épidémies [16], avec une surreprésentation de génotypes EHR [17]. Un phénomène de rétro-contamination du brise-jet peut se produire par projection d’aérosols et de gouttelettes lors de l’utilisation du point d’eau [18]. Le patient peut alors être infecté secondairement à l’exposition à de l’eau contaminée au point d’utilisation [19], vecteur de propagation de PA dans l’environnement (Figure 1). De même, la capacité de PA à former des biofilms est un atout pour coloniser l’environnement hospitalier, qui le rend plus résistant aux antimicrobiens et lui permet de survivre sur de nombreuses surfaces telles que le verre et l’acier, largement utilisés pour les surfaces de soins et la composition de dispositifs médicaux. Les surfaces ainsi contaminées peuvent servir de relais de transmission de PA et engendrer une IAS ou une épidémie, notamment en cas de transmission croisée par un dispositif médical réutilisable contaminé [20] (Figure 1). Parmi les dispositifs particulièrement à risque infectieux, notamment en raison de leur complexité et du risque sous-jacent d’accumulation de souillures organiques (tels que des liquides biologiques), figurent les endoscopes. Plusieurs épidémies à PA ont déjà été rapportées dans les suites de l’utilisation de matériels partagés tels que les gastroscopes, les bronchoscopes ou les cystoscopes comme source d’exposition commune entre les patients [21,22].
La surveillance de la qualité microbiologique de l’eau
Comme évoqué ci-dessus, les patients présentant un profil d’immunodépression (patients en oncohématologie, brûlés…) ou qui sont hospitalisés dans les services de soins intensifs ou de réanimation sont les plus à risque de développer une infection à PA [23]. Dans ces secteurs de soins, la surveillance de la qualité microbiologique de l’eau à la recherche de PA peut être réalisée en routine, selon une stratégie établie par l’équipe de prévention du risque infectieux en fonction de l’épidémiologie locale, du type de patients hospitalisés, de l’architecture et de la maintenance du réseau d’eau [24]. La réalisation de contrôles microbiologiques périodiques de l’eau pour soins standard à la recherche de PA permet de connaître son épidémiologie, qui varie entre services et établissements de soins. En contexte épidémique, souvent lié à la présence d’une source ou d’un réservoir environnemental de PA [25,26], il est recommandé de réaliser des contrôles microbiologiques de l’eau et des surfaces des points d’eau, particulièrement dans les services de soins intensifs et de réanimation [24]. En situation épidémique, les points d’eau à contrôler sont ceux qui exposent potentiellement les patients, soit directement car présents dans leur environnement proche (chambre, salle de bains), soit indirectement par l’exposition des personnels et des matériels de soins (salle de soins, local de désinfection du matériel, office alimentaire…). En cas d’épidémie, une évaluation du risque d’exposition tout au long du parcours de soins des patients impliqués est donc nécessaire pour identifier ces points d’eau et les éventuelles surfaces ou matériels de soins pertinents à prélever.
Recherche dans les eaux de soins
Pour la recherche de PA dans les eaux de soins, il est recommandé de prélever un échantillon d’eau (en premier jet afin d’évaluer la contamination distale du point d’eau et le risque d’exposition direct, ou en second jet afin d’évaluer la contamination globale du réseau d’eau) d’un volume de 250 ml dans un flacon stérile contenant un agent neutralisant des biocides (traditionnellement du thiosulfate de sodium, en concentration finale de 20 mg/L). L’échantillon d’eau ainsi recueilli doit être acheminé rapidement au laboratoire, ou conservé au réfrigérateur pendant une durée maximale de 24 heures. Il est ensuite filtré sur une membrane de porosité de 0,22 μm ensemencée sur un milieu de culture gélosé sélectif des Pseudomonas spp. tel que la gélose au cétrimide, et incubé jusqu’à 72 heures à une température de 30°C en aérobiose (Figure 2). Après incubation, la présence d’une croissance bactérienne est objectivée en dénombrant le nombre d’unités formant colonie (UFC) pour 100 mL d’eau filtrée, et la présence de PA par identification morphologique de colonies pigmentées bleu-vert puis par spectrométrie de masse MALDI-TOF8.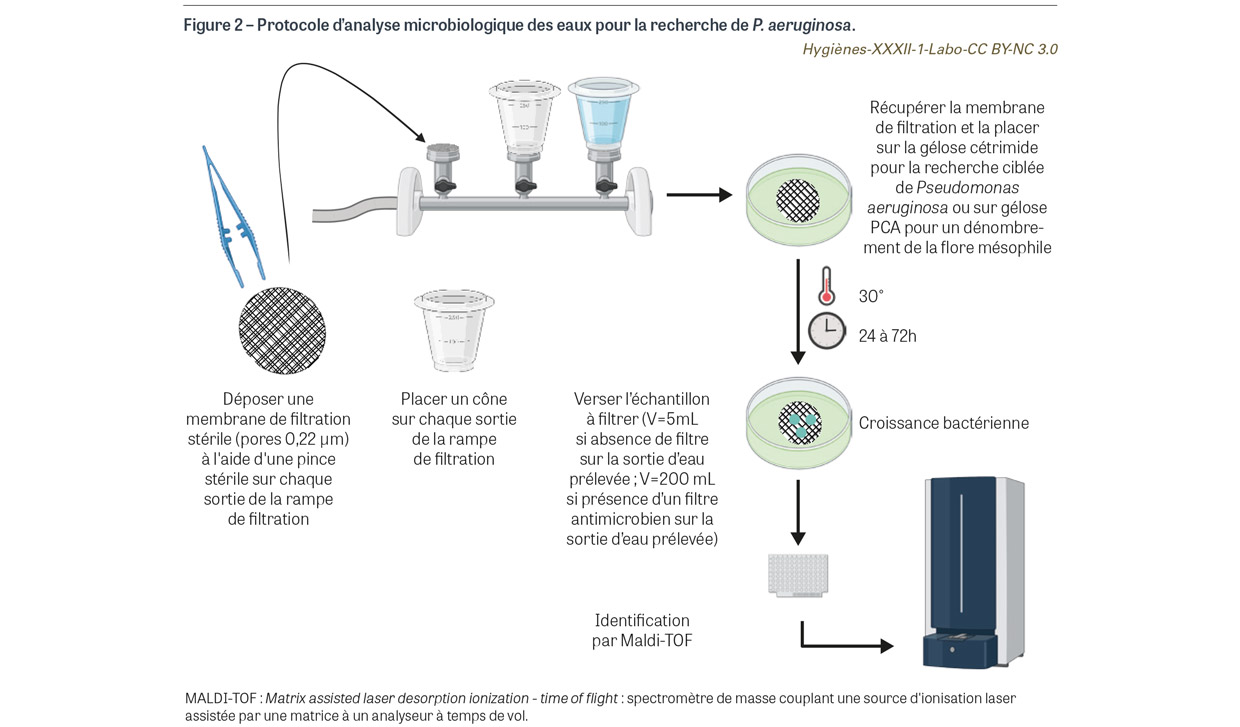
Recherche sur les surfaces de l’environnement hospitalier
Pour la recherche de PA sur les surfaces de l’environnement hospitalier, plusieurs techniques peuvent être utilisées9 : les surfaces peuvent être prélevées à l’aide d’écouvillons stériles ou de lingettes, qui sont ensuite ensemencés sur milieux sélectifs et incubés dans les mêmes conditions que celles citées ci-dessus. Il existe également des géloses « contact » à base de cétrimide sélectives pour PA. Enfin, en cas de phénomène épidémique impliquant un génotype ou un antibiotype particulier de PA, sa détection directe dans l’échantillon par biologie moléculaire (test PCR10 ciblant un gène caractéristique du génotype ou spécifique d’un mécanisme de résistance) peut être envisagée, même s’il n’existe pas à ce jour de méthode standardisée.
Les techniques de typage moléculaire de P. aeruginosa dans le cadre d’investigations
Dans le contexte d’une investigation d’une IAS ou d’une épidémie, l’identification de PA dans des échantillons d’eau ou de surfaces peut conduire à la réalisation d’un typage comparatif entre les souches cliniques et environnementales. Le typage comparatif permet la distinction de sous-groupes d’isolats bactériens (souches) au sein d’une même espèce via la production d’empreintes phénotypiques ou moléculaires spécifiques des isolats [27]. Sur la base des profils établis, il est possible d’apprécier les relations potentielles entre les isolats microbiens. La compréhension de cette parenté phylogénétique est essentielle pour déterminer l’origine et les différentes voies de transmission et de contamination. En effet, en s’appuyant sur les liens épidémiologiques existant entre les isolats, des épidémies peuvent être confirmées ou exclues [28], des souches virulentes ou résistantes peuvent être identifiées et suivies [29] et l’efficacité des mesures de lutte contre l’infection peut être évaluée. À l’heure actuelle, l’intérêt du typage bactérien en santé publique est largement reconnu. Il permet, entre autres, une gestion rationnelle des épidémies via l’allocation efficace de ressources humaines et matérielles, ainsi que la mise en place de programmes cohérents de lutte contre les infections. Différentes méthodes de typage peuvent être utilisées pour caractériser les isolats afin de les comparer les uns aux autres. Les techniques de typage se répartissent généralement en deux grandes catégories : les méthodes non moléculaires (phénotypiques) et les méthodes de typage moléculaires ou génotypiques [30].
Méthodes de typage phénotypiques
Parmi les méthodes de typage phénotypiques les plus couramment utilisées, on peut citer le biotypage, qui consiste à classer les souches notamment selon des profils biochimiques ou morphologiques, ou encore l’antibiotypage qui permet de comparer les profils de résistance aux antimicrobiens, le plus souvent par une méthode de diffusion en milieu gélosé. Le biotypage est rarement utilisé dans le cadre d’un typage épidémiologique car son pouvoir discriminant est faible, notamment en raison de l’occurrence de différents profils biochimiques et morphologiques chez des souches partageant le même génotype. Pareillement, le profil de résistance de PA est soumis à de fortes variations imposées par les pressions de sélection s’appliquant sur la niche. En l’absence de conditions sélectives, certains déterminants de la résistance ne s’expriment pas ou peuvent être perdus en raison de leur localisation plasmidique. Ainsi, cette approche n’est pas idéale lorsqu’il s’agit de relier un réservoir environnemental à un cas clinique puisque les deux niches présentent des caractéristiques différentes, gouvernant le phénotype de résistance exprimé par PA dans chacune des conditions. Le typage par spectrométrie d’absorption infrarouge est une approche phénotypique rapide basée sur l’analyse comparative des composants (principalement glucidiques) de la paroi bactérienne, permettant un dépistage avec une excellente valeur prédictive négative en cas de suspicion de flambée épidémique [31]. Au total, en raison de leur faible reproductibilité, de leur imprécision ou de leur manque de pouvoir discriminant, les méthodes de typage phénotypique, hormis la dernière technique citée, ne sont pas suffisantes pour permettre une différenciation des souches de PA et leur caractérisation à visée épidémiologique.
Méthodes de typage moléculaires ou génotypiques
Les méthodes de typage moléculaires ont largement contribué à l’amélioration des systèmes de surveillance des IAS en permettant de déterminer une éventuelle parenté entre plusieurs isolats de l’espèce PA. Au sein des techniques moléculaires, le typage par technique de rep-PCR (pour repetitive extragenic palyndromic element PCR) s’appuie sur l’amplification de séquences intergéniques répétées, dont la position et le nombre varient d’une souche à l’autre au sein de la même espèce. L’inconvénient majeur de cette technique est son manque de reproductibilité et de standardisation. Aujourd’hui, la technique de rep-PCR est davantage utilisée comme un outil de screening et il convient d’interpréter de manière prudente ses résultats au regard des données épidémiologiques. Durant de longues années, le « gold standard » pour le typage des souches de PA a été l’électrophorèse en champ pulsé (ECP), qui consiste à faire découper l’ensemble du génome bactérien par des enzymes de restriction et à établir, par l’électrophorèse des gros fragments d’ADN11 ainsi générés, un pulsotype révélant la dispersion des sites de restriction au sein de l’ensemble du génome bactérien. Cette technique s’avère assez discriminante mais reste relativement délicate à mettre en œuvre sous l’angle technologique. Plus récemment est apparu le typage par technique multilocus sequence typing12 (MLST) ; il est basé sur le polymorphisme de sept gènes de ménage permettant la détermination d’un profil allélique conduisant in fine à l’attribution d’une séquence type (ST). Le typage MLST de PA, qui s’appuie sur un schéma décrit par Curran et al.13, offre un typage particulièrement robuste et reproductible, permettant une comparaison des isolats à l’échelle internationale, d’où son intérêt certain pour l’étude de la phylogénie des souches de PA [32] dans les études évolutives et les enquêtes épidémiologiques à long terme et à grande échelle (repère spatiotemporel étendu). Ce typage MLST permet l’identification et la surveillance des génotypes EHR. En Europe, les génotypes EHR les plus répandus sont les ST111, ST175, ST235, ST233, ST244, ST277, ST298, ST308, ST357 et ST654 [7]. La démocratisation du séquençage de génomes complets (whole genome sequencing ou WGS) par les techniques de nouvelle génération a permis de développer le typage par la technique du core genome-MLST (cg-MLST) qui repose sur le même principe que le typage MLST et s’appuie sur un schéma élargi afin de sélectionner des milliers de loci (positions) de gènes centraux. Le typage par cg-MLST offre, par rapport à son homologue MLST, un degré de résolution largement supérieur permettant de conduire des enquêtes sur des épidémies qui surviennent dans un repère spatio-temporel étroit à l’échelle hospitalière. Au rythme de l’amélioration des technologies, les méthodes de typage par séquençage WGS deviennent de plus en plus populaires du fait d’un accès facilité et de la baisse de leur coût. Le typage par recherche des polymorphismes nucléotidiques uniques (single nucleotide polymorphism ou SNP), vise à identifier les SNPs au sein d’un panel de génomes en les comparant à une référence unique. L’identification des SNPs offre le pouvoir discriminant le plus élevé par rapport aux autres techniques de typage basées sur le WGS mais il est soumis à d’importantes limitations, et notamment à un manque de standardisation et d’harmonisation du flux de travail, avec, par exemple, l’absence de normalisation des seuils pour définir la parenté des isolats au sein des espèces bactériennes. Au total, pour l’espèce PA, l’approche cg-MLST s’est montrée aussi efficace qu’un typage par SNP pour distinguer les isolats d’une épidémie de ceux non reliés à l’épidémie [33]. Le cg-MLST permet une standardisation des résultats via l’accès public à plusieurs schémas de cg-MLST, servant ainsi de nomenclature internationale normalisée comme base pour la surveillance épidémiologique, et faisant de l’approche cg-MLST la méthode de choix pour le typage des génomes bactériens au sein de l’Union européenne [34]. Une stratégie de typage comparatif à mettre en place dans le cadre d’investigations d’épidémies hospitalières est proposée (Figure 3).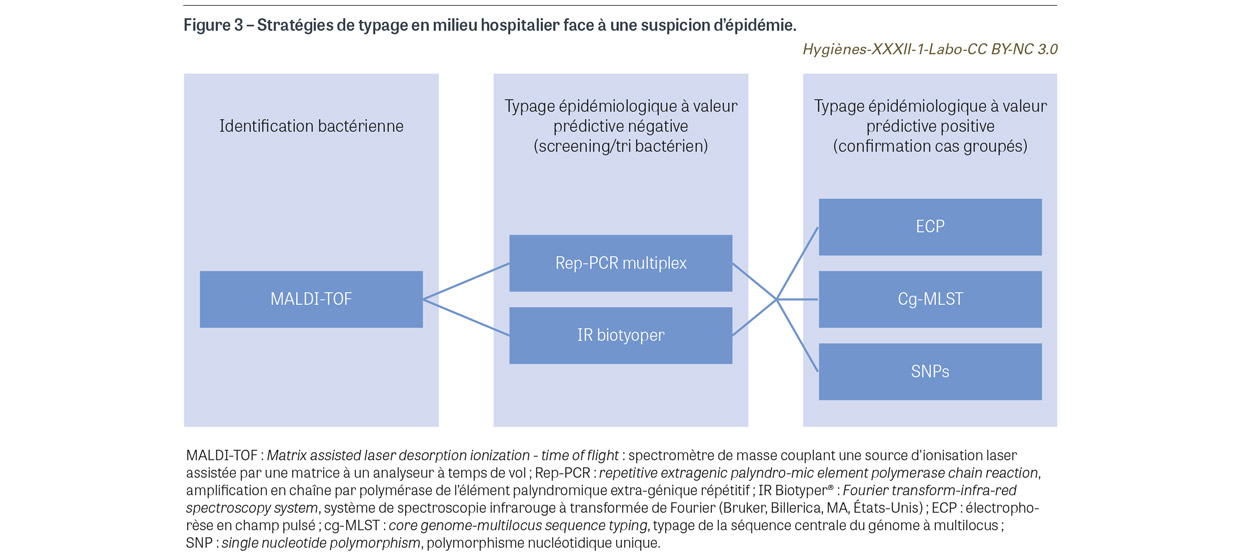
Prévention des infections associées aux soins à Pseudomonas aeruginosa : vers une meilleure gestion de l’environnement
Réservoir environnemental et axes de prévention
L’existence d’un réservoir environnemental compatible avec l’isolat responsable de l’infection suggère le rôle important que l’environnement peut jouer dans la survenue des IAS à PA, même en dehors des épidémies [35]. Les stratégies visant à prévenir et contrôler au mieux ce risque infectieux sont multiples. Elles ont pour principale caractéristique commune de s’appuyer sur deux axes de prévention, d’une part maîtriser la source de contamination, principalement au travers d’une gestion des points d’eau pour lutter contre les réservoirs environnementaux, et d’autre part limiter les voies de transmission, notamment le risque d’infection par transmission croisée qui peut être manuportée ou associée à un dispositif médical partagé contaminé. De manière générale, il est difficile d’évaluer l’impact d’une mesure unique de prévention. En effet, face à une situation de crise, c’est-à-dire en cas d’épidémie, des bundles, correspondant à un ensemble de mesures de prévention, sont mis en place et c’est bien souvent l’association de ces mesures qui permet d’enrayer l’épidémie [16].
Toutes les IAS à PA ne peuvent être prévenues via le contrôle de l’environnement, notamment hydrique, car une part non négligeable des IAS à PA est d’origine endogène, secondaire à une colonisation principalement digestive donc difficilement évitable. Une colonisation respiratoire est également retrouvée chez les patients souffrant de maladie pulmonaire chronique comme la mucoviscidose, la bronchopneumopathie chronique obstructive ou la dilatation des bronches. Néanmoins, la contamination par PA du point d’eau dans la chambre du patient constitue un facteur de risque indépendant de la colonisation digestive par PA [36]. Concernant la part exogène des IAS à PA, celle-ci est variable et dépend de plusieurs éléments comme la qualité microbiologique de l’eau, le respect des mesures de prévention et d’hygiène par le personnel soignant ou encore le type de soins prodigués [37]. La prévention des IAS à PA passe par le contrôle de l’environnement hydrique proche du patient et la mise en place de mesures barrières visant à limiter le risque de transmission croisée.
Qualité de l’eau aux points d’usage : conception des réseaux d’eau et maintenance
La qualité de l’eau aux points d’usage est sous la responsabilité du directeur de l’établissement de santé et doit répondre à des critères microbiologiques stricts, notamment être exempte du pathogène opportuniste PA (niveau cible <1 UFC/100 mL). Afin de s’assurer de la qualité microbiologique aux points d’usage dans les établissements de santé, une veille épidémiologique environnementale par contrôle régulier de ce type de points d’eau est nécessaire. En raison de la multiplicité des points d’eau et de l’impossibilité technique de les prélever tous en termes de ressources humaines et financières, il convient de concevoir un plan d’échantillonnage tenant compte de la structure du réseau d’eau, de la fréquence d’utilisation et des usages, afin d’inclure les points critiques dans l’analyse. Le développement de biofilms dans les réseaux d’eau hospitaliers peut être à l’origine d’un relargage aléatoire d’agrégats microbiens conduisant à une détérioration de la qualité de l’eau. Or la prolifération bactérienne et la formation de biofilms sont favorisées par la stagnation de l’eau ou les dépôts formés par la corrosion [38]. Ainsi, une mesure de base visant à contrôler au mieux l’environnement hydrique et le risque d’IAS à PA nécessite une réflexion en amont sur la conception des réseaux d’eaux afin de supprimer les bras morts et de lutter contre les zones de stagnation de l’eau via un dimensionnement hydraulique adapté des réseaux. Il convient de minimiser le nombre de robinets pour éviter les sorties d’eau sous-utilisées et un faible débit, et de choisir une conception d’évier limitant au maximum le risque d’éclaboussures dues à l’écoulement de l’eau dans les drains [13]. La suppression complète des points d’eau dans les chambres de réanimation est associée à une diminution drastique des IAS [39,40]. De même, la mise en œuvre de soins sans eau est une mesure essentielle pour empêcher l’acquisition de PA à partir de l’eau [41]. Les règles de maintenance des réseaux telles que la maîtrise de la température de l’eau et l’entretien des installations avec la mise en place de traitements anticorrosion et antitartre sont essentielles à la prévention de la prolifération microbienne. Les traitements complémentaires de l’eau avec la sub-chloration de l’eau permettent également de réduire l’inoculum bactérien. La formation de biofilm peut également être limitée par le choix du matériau constitutif du réseau d’eau et la conception des robinets. En raison de ses propriétés bactéricides, le cuivre semble être un matériau de choix pour limiter la formation de biomasse bactérienne. L’utilisation de robinets électroniques à déclenchement automatique sans contact augmente le risque de contamination de l’eau par PA par rapport aux robinets manuels du fait d’une contamination fréquente de l’électrovanne [42]. Dans les secteurs accueillant des patients immunodéprimés, l’eau du réseau doit être filtrée (présence de filtres terminaux antibactériens de porosité 0,22 μm afin de contrôler simultanément le risque lié à Legionella pneumophila et aux bacilles à Gram négatif non fermentant comme PA). Après une phase de validation de l’efficacité des filtres à usage unique, il n’existe pas de réglementation imposant le contrôle microbiologique de l’eau utilisée pour les soins du patient. Cependant, des travaux récents ont montré un risque de colonisation rétrograde de l’effluent, et cela avant la date de péremption du filtre, dans un quart des cas [43]. Les filtres antimicrobiens ne sont pas suffisants pour préserver les patients des pathogènes d’origine hydrique lorsque le réseau d’eau est fortement contaminé [44]. Ainsi, il pourrait être préférable à l’avenir d’associer l’utilisation de filtres antimicrobiens à des prélèvements microbiologiques répétés. Lorsqu’une contamination hydrique par PA est mise en évidence, le remplacement des pommes de douche, siphons ou tuyauteries colonisés ainsi que la désinfection des sorties des points d’eau contaminés peuvent représenter une première mesure barrière. Néanmoins, bien souvent, ces mesures sont insuffisantes pour l’éradication de PA. Il est alors possible de procéder à des chocs chimiques ou thermiques sur le réseau d’eau et de placer des filtres antimicrobiens sur toutes les sorties d’eau. Cependant, l’utilisation répétée de biocides et de désinfectants n’est pas sans conséquence sur l’intégrité du réseau d’eau, sur l’émergence de nouvelles résistances et sur l’écotoxicité d’aval [45].
Suppression des points d’eau et réalisation des soins « sans eau » : la solution
Actuellement, la suppression des points d’eau en association avec la réalisation de soins « sans eau », notamment dans les services à risque tels que les unités de soins intensifs et les réanimations, semble offrir la solution la plus durable et la plus efficace pour prévenir les infections à PA à point de départ hydrique. En effet, Schärer et al. [46] ont mis en évidence une nouvelle colonisation des siphons d’évier par une souche de PA multirésistante aux antibiotiques à caractère épidémique seulement trois semaines après leur remplacement. Les siphons sont reconnus comme un point critique de colonisation et de persistance pour PA mais également de propagation de l’antibiorésistance [47]. L’éradication de PA au sein de cette niche semble illusoire et, par conséquent, la présence de lavabos dans ces secteurs de soins expose à un risque d’épidémie à PA et à d’autres pathogènes opportunistes tels que les OPPPs ou les Enterobacterales, notamment celles qui sont productrices de carbapénémases [40,46]. La principale barrière à la suppression des points d’eau est d’ordre organisationnel puisqu’elle nécessite de revoir complètement le déroulement des soins infirmiers. Il semble essentiel qu’une réflexion en amont de cette suppression soit menée afin de proposer une nouvelle organisation compatible avec les contraintes du terrain et d’éviter un bouleversement des pratiques.
P. aeruginosa, un pathogène opportuniste à expression nosocomiale
Au-delà de sa participation à l’éradication des réservoirs environnementaux à PA, la suppression des points d’eau en unités de soins intensifs et en réanimation est associée à une réduction significative du taux de colonisation des patients à PA, réduction d’autant plus marquée que la durée du séjour est longue [39]. Cette observation est majeure, sachant qu’une part non négligeable des IAS à PA est d’origine endogène en raison d’un portage infra-détectable, l’infection se déclarant par pression de sélection hospitalière, notamment via les antibiotiques. Même si PA n’est pas une bactérie commensale des microbiotes humains, 1,5% à 8,2% des adultes sains, selon les études [48,49], sont décrits comme colonisés au niveau digestif par PA, avec une fréquence plus élevée, de l’ordre de 11,6% à 43% [50,51], chez les patients admis en réanimation en raison de pathologies sous-jacentes ou de situations particulières prédisposant à une colonisation au long cours à PA. La colonisation digestive à PA peut atteindre des taux particulièrement élevés au cours d’une hospitalisation aiguë (57,8%) ou en cas de séjour de longue durée (51,6%) [52]. Le risque de portage de PA augmente au cours d’une hospitalisation, notamment lors de l’utilisation d’antibiotiques inactifs contre PA [36] exposant à un risque de dysbiose favorable à l’implantation et à la colonisation digestive par PA [36,50]. On ne peut pas exclure que ces IAS à PA trouvent leur point de départ dans une exposition communautaire à l’origine d’un portage transitoire qui, à la faveur de pressions de sélection lors d’une hospitalisation, se traduit par une infection. Il s’agit là du concept de pathobiome hydrique associé aux soins, où finalement la survenue d’une IAS est le fruit d’un profil de patient particulier (immunodéprimé par exemple), d’un type de soins (invasifs par exemple) ou d’une pression de sélection (telle qu’une antibiothérapie large spectre) et d’une exposition environnementale. Cette hypothèse est étayée par la prévalence croissante du portage intestinal de PA, estimée à environ 10% chez les patients hospitalisés en réanimation [53] et jusqu’à 51% chez les résidents des établissements de soins de longue durée [52]. Le portage intestinal de PA au sein de la population hospitalière est largement méconnu et sous-estimé puisqu’actuellement les tests de dépistage de colonisation rectale par PA ne sont pas effectués dans les hôpitaux. Le dépistage des bactéries multirésistantes étant réalisé sur des milieux sélectifs ciblant les bactéries hautement résistantes aux antibiotiques, seuls les phénotypes de PA résistants aux carbapénèmes sont identifiés de manière fortuite, ce qui conduit à une sous-estimation du portage communautaire de PA.
Conclusion
P. aeruginosa constitue un risque croissant d’infections graves chez les patients fragiles hospitalisés en réanimation ou en soins intensifs. L’origine des contaminations peut être endogène, favorisée par la pression antibiotique à partir d’une colonisation intestinale infraclinique, ou exogène à partir de l’environnement hospitalier (mains du personnel, matériels, environnement hydrique), ou mixte. La survenue de cas groupés ou d’épidémies intra-hospitalières doit conduire à des investigations microbiologiques poussées destinées à identifier une cause exogène possible à partir des souches de patients et de l’environnement ; à cet égard, les techniques génotypiques de cg-MLST ou éventuellement de SNP sont recommandées (Figure 4). Pour prévenir et maîtriser les contaminations hydriques, les mesures destinées à réduire la contamination des réseaux d’eau s’avèrent toutes décevantes, ce qui conduit de plus en plus à préconiser des chambres sans point d’eau dans les services à risque ; cette stratégie exige en corollaire une réorganisation des soins paramédicaux. Enfin, la capacité des souches de PA à développer des résistances aux antibiotiques et aux biocides en général constitue un autre élément majeur de préoccupation qui justifie la mise en œuvre de l’ensemble des mesures préventives et curatives détaillées dans cette synthèse, avec la nécessité de proposer des stratégies institutionnelles pour tendre à maîtriser le risque.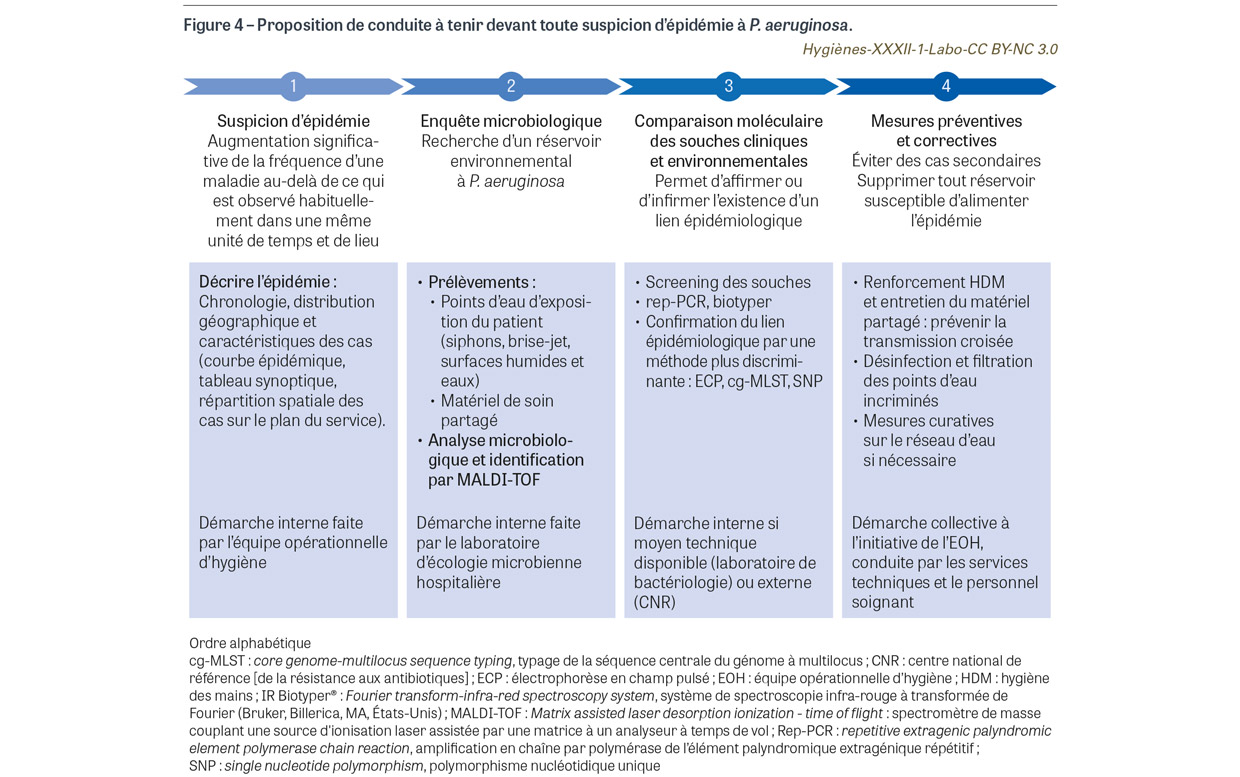
Notes :
1- Réanimation-Réseau d’alerte, d’investigation et de surveillance des infections nosocomiales.
2- Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa, et Enterobacter spp.
3- World Health Organization (WHO). WHO Bacterial Priority Pathogens List, 2024: bacterial pathogens of public health importance to guide research, development and strategies to prevent and control antimicrobial resistance. Geneva: WHO; 2024. 72 p. Accessible à : https://iris.who.int/bitstream/handle/10665/376776/9789240093461-eng.pdf?sequence=1 (Consulté le 20-06-2024).
4- Epidemic high-risk.
5- Centre national de référence de la résistance aux antibiotiques. Rapport annuel d’activité 2023, année d’exercice 2022. Saint-Maurice: Santé publique France; 2023. 163 p. Accessible à : https://www.cnr-resistance-antibiotiques.fr/ressources/pages/CNR_bilan_2022.pdf (Consulté le 18-06-2024).
6- Verona integron-encoded metallo-β-lactamase 2.
7- New Delhi metallo-β-lactamase.
8- Matrix assisted laser desorption ionization - time of flight : spectromètre de masse couplant une source d'ionisation laser assistée par une matrice à un analyseur à temps de vol.
9- Voir Romano Bertrand S, Pozzetto B, Decousser JW. Les prélèvements de surface pour la recherche ciblée de micro-organismes spécifiques : intérêts et limites. Hygienes 2023;31(1);63-69.
10- Polymerase chain reaction, amplification en chaîne par polymérase.
11- Acide désoxyribonucléique.
12- Typage de séquences multilocus.
13- Curran B, Jonas D, Grundmann H, et al. Development of a multilocus sequence typing scheme for the opportunistic pathogen Pseudomonas aeruginosa. J Clin Microbiol. 2004;42(12):5644-5649. Et : Public databases for molecular typing and microbial genome diversity. Pseudomonas aeruginosa [Internet]. Oxford (UK): University of Oxford; 2018. Accessible à : https://pubmlst.org/organisms/pseudomonas-aeruginosa/ (Consulté le 18-06-2024).